تنوع ژنتیکی ماهی تیلاپیا نیل ( Oreochromis niloticus ) در سراسر آفریقای غربی
خلاصه
تیلاپیا نیل ( Oreochromis niloticus) یک گونه آبزی پروری در سطح جهانی است که به سرعت به عنوان یک کالای پرورشی وضعیت می یابد. در غرب آفریقا، منابع ژنتیکی تیلاپیا نیل وحشی فراوان است اما دانش ساختار جمعیتی در مقیاس خوب و الگوهای تنوع ژنتیکی طبیعی محدود است. همزمان با این، رشد فزاینده ای در آبزی پروری تیلاپیا در غنا و سایر کشورهای منطقه است که زیربنای منابع ژنتیکی در دسترس محلی است. این مطالعه با استفاده از 192 نشانگر پلی مورفیسم تک نوکلئوتیدی (SNP) یک بررسی ژنتیکی از تیلاپیا نیل در سراسر آفریقای غربی انجام داد و از 23 جمعیت وحشی در هشت کشور (بنین، بورکینافاسو، ساحل عاج، غنا، توگو، مالی، گامبیا و سنگال) نمونه برداری کرد. ، نشان دهنده حوضه های آبریز اصلی حوضه های رودخانه ولتا، نیجر، سنگال و گامبیا است.F ST = 0.144)، که تا حد زیادی مربوط به حوضه های رودخانه های اصلی و، تا حدی، حوضه های فرعی است. دو جمعیت از رودخانه گامبیا (Kudang و Walekounda)، یکی از رودخانه نیجر غربی (دریاچه Sélingué) و یک جمعیت از رودخانه Red Volta بالایی (Kongoussi) سطوح پایینتری از تنوع و تمایز ژنتیکی بالا را در مقایسه با سایر جمعیتها نشان دادند. جمعیت های جدا شده ژنتیکی در سراسر منطقه وجود دارد. ساختار ژنتیکی در حوضه ولتا همیشه از الگوی مورد انتظار برای حوضه های زیر رودخانه پیروی نمی کند. این مطالعه ساختار و تمایز ژنتیکی واضحی را در میان جمعیتهای تیلاپیا نیل غرب آفریقا که با الگوهای گسترده یافت شده در مطالعات قبلی مطابقت دارد، شناسایی میکند. علاوه بر این، ما شواهد جدیدی برای ساختار ژنتیکی در مقیاس ریز در حوضه ولتا و تفاوتهای ژنتیکی قبلاً ناشناخته جمعیتها در گامبیا ارائه میکنیم. مجموعه نشانگر 192 SNP مورد استفاده در این مطالعه ابزار مفیدی برای افتراق جمعیت ماهی تیلاپیا است و توصیه می کنیم این مجموعه نشانگر را در غربالگری جمعیت آتی بگنجانید.O. niloticus . نتایج ما اساس یک پلت فرم محکم برای تحقیقات آینده در مورد منابع ژنتیکی تیلاپیا وحشی در غرب آفریقا، و شناسایی ژرم پلاسم بالقوه ارزشمند برای استفاده در برنامههای اصلاحی در حال انجام برای آبزی پروری را تشکیل میدهد.
معرفی
اورئوکرومین ها زیر خانواده ای از خانواده ماهی های سیکلید هستند که بومی آفریقا و بخش هایی از شام هستند که اهمیت تجاری زیادی برای آبزی پروری و صید ماهی دارند . تیلاپیا نیل، Oreochromis niloticus ، دارای توزیع طبیعی گسترده ای است که از حوضه رودخانه نیل به سمت جنوب از طریق دریاچه های دره ریفت شرقی و غربی در شرق آفریقا، و از غرب از طریق حوضه های دریاچه چاد، نیجر، بنوه، ولتا، گامبیا و رودخانه های سنگال گسترش می یابد. 1 . این گونه از دیدگاه طبقه بندی به خوبی مورد مطالعه قرار گرفته است. ساختار زیرگونه پیچیده و توزیع O. niloticusمستند شده است، به ویژه در سراسر شرق آفریقا که در آن شبکه ای از دریاچه های متنوع جغرافیایی و بیولوژیکی تکامل آن را شکل داده اند. هفت زیر گونه O. niloticus بر اساس روش های طبقه بندی سنتی شناسایی شده است. همه به جز یکی – O. niloticus niloticus – در بین دریاچه های دره ریفت در شرق آفریقا توزیع شده اند .
مطالعات مولکولی تمایز واضحی را بین جمعیتهای آفریقای غربی و شرقی O. niloticus شناسایی کرده است . جمعیت دره و (iii) زهکشی نیل و جمعیت دره ریفت کنیا 5 ، 6 . یک الگوی سلسله مراتبی از تمایز ژنتیکی در O. niloticus مشاهده شده است، به موجب آن تأثیرات رویدادهای دیرینه-جغرافیایی و اقلیمی اصلی عامل غالب در مقیاس کلان جغرافیایی است و سیستم های رودخانه ای امروزی بر اتصال ژنتیکی و الگوهای ساختار ژنتیکی در مقیاس های جغرافیایی کوچکتر تأثیرگذار هستند . همان نویسندگان همچنین پیشنهاد کردهاند که قابلیتهای پراکندگی محدود همراه با جفتگیری غیر تصادفی ناشی از رفتارهای اجتماعی نیز ممکن است بر ساختار جمعیت در مقیاس کوچک تأثیر بگذارد. مطالعات دیگر نشان می دهد که جمعیت های O. niloticus جدا شده و قبلاً توصیف نشده در کنیا می توانند تمایز ژنتیکی قوی را از سایر جمعیت های مجاور نشان دهند و ممکن است منابع ژنتیکی وحشی جدیدی را با اهمیت بالقوه برای بهره برداری در فعالیت های تجاری آبزی پروری تشکیل دهند .. علاوه بر این، مطالعات الگوهای تنوع DNA میتوکندری پیچیدگیهای بیشتری را در میان جمعیتهای O. niloticus نشان دادهاند ، با شواهدی از هیبریداسیون تاریخی بین گونههای O. aureus 8 و O. leucosticus 9 که به ترتیب در مناطق غربی و شرقی آفریقا رخ میدهند.
اگرچه توجه زیادی به خصوصیات جمعیت و ساختار ژنتیکی O. niloticus در شرق آفریقا شده است، مطالعات نسبتا کمی بر روی جمعیت های سراسر غرب آفریقا با وضوح بالا متمرکز شده است. این در حالی است که غرب آفریقا بزرگترین منطقه ایکتیوری است که به طور طبیعی توسط O. niloticus اشغال شده است. مطالعات قبلی تمایز ژنتیکی را در مقیاس های وسیع (یعنی 1000 کیلومتر کیلومتر) نشان داده اند 3 ، 4 ، 5 . با این حال، پوشش فضایی این مطالعات محدود است و در نتیجه دانش الگوهای مقیاس دقیق تر ساختار ژنتیکی O. niloticus در غرب آفریقا نامشخص است.
Oreochromis niloticus تا حد زیادی برجسته ترین گونه ماهی تیلاپیا است که توسط آبزی پروری تولید می شود و پرورشی ترین گونه ماهی گرمسیری در سطح جهان است. تقریباً 4.1 میلیون تن تیلاپیا نیل توسط آبزی پروری در سال 2017 تولید شد که ارزش آن 7.6 میلیارد دلار آمریکا بود . جمعیت های O. niloticus آفریقای غربی سهم قابل توجهی در توسعه منابع ژنتیکی پرورشی داشته اند که زیربنای بسیاری از تولیدات جهانی است. برای مثال، حداقل چهار مورد از هشت جمعیت پایهگذار (و احتمالاً پنج جمعیت) از سویه بسیار موفق تیلاپیا (GIFT) از O. niloticus یا به طور مستقیم از جمعیتهای وحشی در غنا و سنگال یا از جمعیتهای کشتشده به دست آمده بودند. سهام با منشاء غنا11 . سویه GIFT از اوایل دهه 1990 تحت پرورش انتخابی مستمر در آسیای جنوب شرقی قرار گرفته است و به طور گسترده به عنوان تأثیر قابل توجهی بر توسعه آبزی پروری تیلاپیا در سراسر منطقه شناخته شده است 12 ، 13 . علاوه بر این، یک برنامه اصلاحی انتخابی مبتنی بر O. niloticus وحشی که منبع محلی است، به سرعت توسط کشاورزان آبزی پروری در غنا محبوبیت پیدا می کند و مزایای اقتصادی و افزایش بهره وری اولیه را برای کشور به ارمغان می آورد 14 ، 15 ، 16 . چنین موفقیت هایی نشان دهنده ارزش بالقوه ای است که می توان از این منابع ژنتیکی وحشی به دست آورد، مشروط بر اینکه رویکردهای مدیریتی و فناوری مناسب اعمال شود.
آبزی پروری ماهی تیلاپیا در سال های آینده برای رشد سریع در سراسر جنوب صحرای آفریقا آماده است. با افزایش آبزی پروری تیلاپیا در سراسر جنوب صحرای آفریقا، انگیزه ای برای درک بهتر توزیع منابع ژنتیکی وحشی و الگوهای ساختار جمعیت در سراسر توزیع طبیعی O. niloticus به طور مشابه افزایش می یابد . محبوبیت تاریخی ماهی تیلاپیا نیل به عنوان ماهیگیری صید و برای تولید آبزی پروری شاهد جابجایی مکرر آن و معرفی عمدی آن به بسیاری از مکانهایی است که در ابتدا یافت نشد. منشأ ژنتیکی چنین معرفیهایی اغلب مبهم یا ناشناخته است، اما ممکن است تأثیر مهمی بر ترکیب ژنتیکی جمعیتهای وحشی داشته باشد.
مزایای بررسی ویژگی های ژنتیکی مولکولی ماهی تیلاپیا نیل وحشی دو برابر است. در مرحله اول، درک ساختار جمعیت وحشی می تواند به مدیریت یا حفاظت از منابع ژنتیکی وحشی، به ویژه در زمینه اثرات بالقوه ناشی از فرار آبزی پروری کمک کند. ثانیاً، شناسایی جمعیتهای وحشی از نظر ژنتیکی متمایز و متنوع، مزایای بالقوهای برای برنامههای اصلاح نژاد آبزی پروری دارد، که به تنوع ژنتیکی گسترده در جمعیتهای بنیانگذار آنها برای دستیابی به بهبود بهرهوری پایدار و اجتناب از مشکلات تجمع همخونی نیاز دارد. با ارائه بینش در مورد مدیریت و استفاده از منابع ژنتیکی تیلاپیا، داده های ژنتیکی مولکولی درک بهتری را در شرایطی ارائه می دهد که توسعه آبزی پروری در مقیاس بزرگ در غرب آفریقا در حال رشد است و استفاده صحیح از منابع بسیار مهم است.
این مطالعه فرصتی را برای درک الگوهای ساختار ژنتیکی و عوامل مؤثر بر تنوع در سراسر منطقه، مستندسازی بهتر منابع ژنتیکی طبیعی تیلاپیا نیل در غرب آفریقا، و ایجاد بیشتر بر روی تحقیقات قبلی برای درک عوامل بالقوه کلان یا خرد جغرافیایی مؤثر بر تنوع آنها ارائه میکند. . با استفاده از نشانگرهای پلی مورفیسم تک نوکلئوتیدی (SNP)، ما 23 جمعیت را در پنج حوضه رودخانه در غرب آفریقا برای ارزیابی الگوهای فضایی تنوع ژنتیکی و تمایز ژنتیکی جمعیتهای تیلاپیا وحشی بومی نیل مطالعه کردیم. اهداف خاص این مطالعه عبارتند از: (1) مشخص کردن تنوع ژنتیکی جمعیت تیلاپیا نیل در سراسر غرب آفریقا با استفاده از نشانگرهای SNP. (2) کمیت تفاوت های نسبی ساختار ژنتیکی در جمعیت تیلاپیا نیل در مقابل حوضه های آبخیز در سراسر غرب آفریقا. (3) ویژگیهای ژنتیکی جمعیتهای تیلاپیا نیل را که بهعنوان بخشی از توزیع طبیعی آن شناخته نشدهاند، اما بهطور محلی به عنوان منابع این گونه شناخته شدهاند، کاوش کنید. و (4) شناسایی عوامل بالقوه موثر در الگوهای تنوع ژنتیکی و تمایز در جمعیت تیلاپیا نیل غرب آفریقا. ما پیشبینی میکنیم که این بینش روشنتری نسبت به جمعیتهایی که ممکن است دارای ارزش بالقوه برای حفاظت یا استفاده بیشتر در برنامههای توسعه آبزی پروری در حال انجام برای منطقه باشند، ارائه دهد. و (4) شناسایی عوامل بالقوه موثر در الگوهای تنوع ژنتیکی و تمایز در جمعیت تیلاپیا نیل غرب آفریقا. ما پیشبینی میکنیم که این بینش روشنتری نسبت به جمعیتهایی که ممکن است دارای ارزش بالقوه برای حفاظت یا استفاده بیشتر در برنامههای توسعه آبزی پروری در حال انجام برای منطقه باشند، ارائه دهد. و (4) شناسایی عوامل بالقوه موثر در الگوهای تنوع ژنتیکی و تمایز در جمعیت تیلاپیا نیل غرب آفریقا. ما پیشبینی میکنیم که این بینش روشنتری نسبت به جمعیتهایی که ممکن است دارای ارزش بالقوه برای حفاظت یا استفاده بیشتر در برنامههای توسعه آبزی پروری در حال انجام برای منطقه باشند، ارائه دهد.
مواد و روش ها
منشاء و محل جمعیت نمونه گیری
بیست و سه جمعیت O. niloticus وحشی از هشت کشور (بنین، بورکینافاسو، ساحل عاج، گامبیا، غنا، مالی، سنگال و توگو) غرب آفریقا نمونه برداری شدند که نشان دهنده حوضه های آبریز اصلی ولتا، نیجر، سنگال و حوضه های رودخانه گامبیا (شکل 1 ). الگوهای تمایز ژنتیکی در مقیاس ظریف در حوضه ولتا مورد هدف قرار گرفتند. دو تا پنج نمونه جمعیت از چندین زیرحوضه، از جمله دریاچه ولتا، مخزن بزرگی که توسط سد آکوسومبو در سال 1965 ساخته شد، و هر یک از چهار شاخه اصلی آن، یعنی رودخانه سیاه ولتا (همچنین به عنوان موهون شناخته میشود) گرفته شد. ، رودخانه های قرمز (نازینون) و ولتا سفید (ناکامبه) و رودخانه اوتی (جدول 1 )). یک سایت از حوضه کومو (بورکینافاسو) و رودخانه کلوکپا (غنا) نیز نمونهبرداری شدند که به عنوان بخشی از توزیع طبیعی O. niloticus 1 شناخته نمیشوند، اما منابع شناختهشده گونهها به صورت محلی هستند.
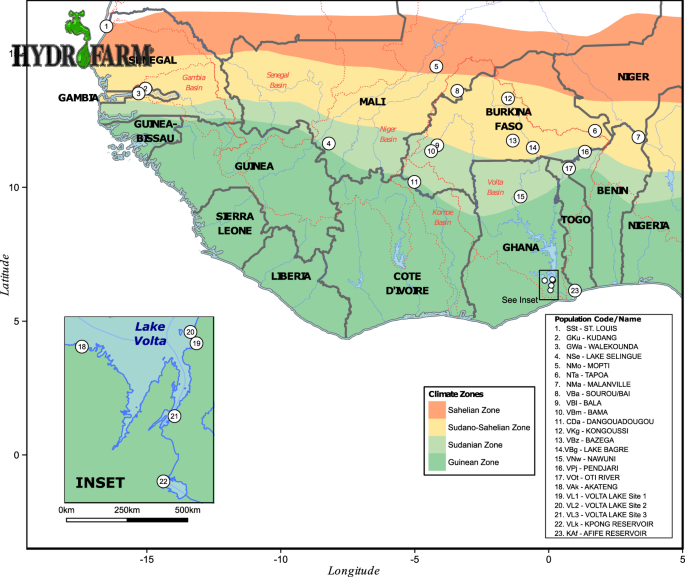
جدول 1 مکان، حوضه اصلی رودخانه و تاریخ جمع آوری جمعیت های تیلاپیا نیل وحشی نمونه برداری شده در سراسر آفریقای غربی.جدول اندازه کامل
مجموعه نمونه
در مجموع 1001 ماهی منفرد از سال 2009 تا 2011 نمونه برداری شد. ماهی ها به طور مستقیم از قایق های ماهیگیری تجاری یا در مکان های فرود مناطق ماهیگیری شناخته شده جمع آوری شدند. یک باله دمی تقریباً 1 × 2 سانتی متر با استفاده از پنس و یک جفت قیچی تمیز و تیز بریده شد و در ویال های برچسب دار جداگانه با استفاده از اتانول 95 درصد نگهداری شد. تمام نمونه ها تا زمان پردازش بعدی در دمای اتاق نگهداری شدند. هر سایت نمونه با استفاده از یک گیرنده GPS دستی ارجاع جغرافیایی شد. بسته به در دسترس بودن، بین 24 تا 50 نفر از هر سایت جمع آوری شد. جنسيت هر ماهي از آزمايش خارجي در هنگام نمونهبرداري نامشخص بود، و فرض بر اين است كه نسبت جنسيت يكنواختي از نر به ماده نمونهبرداري شده است. خلاصه ای از هر مکان نمونه برداری، شامل جزئیات مرجع جغرافیایی، در جدول 1 توضیح داده شده است.
بیانیه اخلاق
حیوانات آزمایشی زنده برای این مطالعه استفاده نشد. بنابراین الزام برای تایید اخلاق حیوانات قابل اجرا نبود. از ماهیهای مرده صید شده توسط ماهیگیران تجاری در مقیاس کوچک با استفاده از تجهیزات استاندارد و روشهای معمول، از ماهیهای مرده گرفته شد. ماهیگیران توسط محققان این مطالعه سفارش داده نشدند و حیوانات مورد مطالعه در روز صید به طور کامل خریداری یا در محل فروش به عموم نمونهبرداری شدند.
پروتکل استخراج DNA
پس از جمع آوری، استخراج DNA با استفاده از کیت تجاری موجود (Promega WIZARD® Genomic DNA Purification Kit) طبق دستورالعمل سازنده انجام شد و تا پردازش بعدی در دمای 20- درجه سانتیگراد نگهداری شد. استخراج DNA در آزمایشگاه مرکز بین المللی تحقیق و توسعه دام در مناطق نیمه مرطوب (CIRDES)، بورکینافاسو انجام شد. نمونه ها با غلظت DNA 50 نانوگرم در 1 استاندارد شدند و با یخ خشک از طریق هوا به دانشگاه واگنینگن برای ژنوتیپ فرستاده شدند.
ژنوتیپ پلی مورفیسم تک نوکلئوتیدی (SNP).
ژنوتیپ های چند جایگاهی از 192 نشانگر SNP، انتخاب شده از مجموعه ای از 384 نشانگر SNP تولید شده برای O. niloticus 18 تولید شدند. ژنوتیپ در یک روش مالتی پلکس 192 SNP با استفاده از روش گلدن گیت (Illumina) انجام شد و بر روی یک پلت فرم BeadXpress با استفاده از فناوری Veracode مستقر شد. در مجموع 15 SNP در سنجش روی کروموزوم ها/گروه های پیوندی قرار گرفتند که در آن ها ارتباط های شناخته شده ای با تعیین جنسیت (LG1، LG20، LG23) در تیلاپیا نیل وجود دارد 19 ، 20با این حال، این SNPهای مورد استفاده در مجاورت این مناطق مرتبط با جنسیت قرار نداشتند (دادهها نشان داده نشده است) و بنابراین کمترین خطر را برای سوگیری نتایج مطالعه دارند. حداقل نرخ تماس 0.8 برای امتیازدهی ژنوتیپ ها استفاده شد. فهرست کاملی از نشانگرهای SNP مورد استفاده، گروه پیوند طبق گویون و همکاران . 21 و شماره دسترسی dbSNP هر SNP در بخش مواد تکمیلی آورده شده است.
تحلیل های آماری
تنوع ژنتیکی
تعداد جایگاه های چندشکلی، هتروزیگوسیتی مشاهده شده و هتروزیگوسیتی مورد انتظار برای هر جمعیت با استفاده از بسته R adegenet 22 محاسبه شد. غنای آللی هر جمعیت با استفاده از بسته R hierfstat 23 بر اساس حداقل حجم نمونه 15 نفر محاسبه شد که تفاوت در اندازه نمونه و شکست ژنوتیپ را نشان می دهد. آزمونهای تعادل هاردی واینبرگ (HWE) در هر مکان برای هر جمعیت با استفاده از تابع HWE.test.genind() adegenet ، بر اساس آماره مجذور کای انجام شد. مقادیر P هر آزمون با استفاده از روش نرخ کشف نادرست بنجامینی و هوچبرگ 24 در جامعه تنظیم شد.تابع p.adjust() در R 25 ، به سطح معنی داری معادل 0.05 = α.
اندازه موثر جمعیت ( Ne )
اندازه جمعیت موثر و فاصله اطمینان 95 درصد آن برای هر جمعیت با استفاده از روش عدم تعادل پیوندی Waples 26 ، همانطور که در NeESTIMATOR نسخه 2.01 27 اجرا شده است، برآورد شد . یک مدل جفت گیری تصادفی با استفاده از P CRIT (کمترین فرکانس آللی) 0.05 استفاده شد.
تمایز جمعیت و فواصل ژنتیکی
آمار F رایت ( F ST ، F IS و F IT ) با استفاده از ARLECORE، نسخه کنسول ARLEQUIN v3.5 28 ، برای تعیین نسبت تنوع ژنتیکی تقسیمبندی شده در داخل و بین سطوح مختلف زیرساختار جمعیت (یعنی فردی ) برآورد شد. ، زیر جمعیت و کل جمعیت). F ST دو به دو بین جمعیت ها نیز با استفاده از ARLECORE محاسبه شد، با اهمیت مقادیر دوتایی با استفاده از رویکرد مونت کارلو غیر پارامتری (1000 جایگشت) آزمایش شد. یک تصحیح بونفرونی با استفاده از تابع p.adjust() در R 25 به مقادیر P اعمال شد.برای کنترل احتمال خطاهای نوع I مرتبط با مقایسه های متعدد. برای بررسی اهمیت عوامل اصلی جغرافیایی مانند حوضه های رودخانه (حوضه های آبخیز) بر ساختار ژنتیکی در ماهی تیلاپیا نیل، تجزیه و تحلیل سلسله مراتبی تغییرات مولکولی (AMOVA) با استفاده از ARLEQUIN اجرا شد. چندین مدل بر اساس گروهبندیهای مختلف جمعیتها آزمایش شدند که محرکهای توضیحی منطقی تمایز ژنتیکی را نشان میدهند. هدف در اینجا کشف این است که چه گروههایی میتوانند تنوع ژنتیکی مشاهدهشده را بهتر توضیح دهند وقتی بین گروهها ( F CT )، بین جمعیتهای درون گروهی ( F SC ) و بین جمعیتها ( F ST ) تقسیم شوند.). جمعیت توسط (1) حوضه رودخانه اصلی گروه بندی شدند. (2) حوضه ها و حوضه های فرعی رودخانه های اصلی (در صورت وجود). و (3) مناطق آب و هوایی.
جداسازی با تست فاصله
برای آزمایش اینکه آیا الگوهای تمایز ژنتیکی از یک مدل جداسازی به فاصله از واگرایی جمعیت پیروی می کنند، آزمون Mantel برای همبستگی بین فواصل جغرافیایی (km) و فواصل ژنتیکی ( F ST ) بر اساس 10000 جایگشت با استفاده از تابع mantel.randtest() انجام شد. در adegenet . فواصل جغرافیایی استفاده شده، فواصل خط مستقیم بین مکانهای نمونه جمعیتی است که از مختصات طول و عرض جغرافیایی با استفاده از تابع earth.dist() فسیل R بسته 29 محاسبه میشود .
تجزیه و تحلیل اجزای اصلی فضایی (sPCA)
الگوهای فضایی تنوع ژنتیکی در O. niloticus بیشتر از طریق تجزیه و تحلیل اجزای اصلی فضایی (sPCA) فراوانیهای آللی جمعیت مورد بررسی قرار گرفت . این روش امکان تجزیه و تحلیل تنوع در فراوانی های آللی و خودهمبستگی فضایی را در بین جمعیت ها فراهم می کند. sPCA با استفاده از تابع spca() adegenet 22 انجام شد. اطلاعات مکانی به تجزیه و تحلیل بر اساس یک شبکه اتصال نمودار گابریل از سایت های نمونه جمعیت گنجانیده شد. تستهای مونت کارلو برای وجود ساختار فضایی جهانی و محلی با استفاده از توابع global.rtest () و local.rtest() adegenet انجام شد.به ترتیب بر اساس 9999 جایگشت. در اینجا، ساختار جهانی به همبستگی فضایی مثبت (یعنی جمعیتهای نزدیکتر به یکدیگر شبیهتر هستند) و ساختار محلی به خود همبستگی فضایی منفی اشاره دارد (جمعیتهای نزدیکتر متفاوتتر هستند). به دلیل سوگیریهای نمونهگیری بالقوه در فرکانسهای آللی ناشی از وجود انواع نادر، مجموعه دادهها برای استفاده از مکانهایی که فرکانس آللی جزئی (MAF) بیشتر از 0.01 در همه افراد داشتند، کوتاه شد.
خوشه بندی ژنتیکی – رویکرد بیزی
رویکرد خوشهبندی بیزی اجرا شده در نرمافزار STRUCTURE v2.3 برای تقسیمبندی دادههای ژنوتیپ بر اساس تعداد از پیش تعریفشده گروهها ( K ) که با انتظارات تعادل هاردی-واینبرگ و تعادل پیوندی بین جایگاههای 31 و 32 مطابقت دارد، استفاده شد. یک مدل ترکیبی با فرکانسهای آللی همبسته برای تخمین احتمال عضویت در گروه ( Q ) برای هر فرد برای K = 2 تا K = 20 انتخاب شد . دوره سوختگی 150000 تکرار. پارامترهای پیش فرض در غیر این صورت استفاده شد. برای شناسایی محتملترین تعداد خوشهها، روش Evanno 33 استبا استفاده از SRUCTURE HARVESTER 34 اجرا شد. CLUMPP 35 برای تراز کردن خوشه ها در سراسر اجراهای تکراری و محاسبه میانگین تخمین Q برای هر فرد استفاده شد که با استفاده از DISTRUCT 36 تجسم شد .
خوشه بندی ژنتیکی – رویکرد چند متغیره
یک رویکرد جایگزین برای شناسایی خوشه های ژنتیکی متمایز از طریق تجزیه و تحلیل متمایز اجزای اصلی (DAPC) 37 است. DAPC متغیرهای مصنوعی (عملکردهای متمایز) را ایجاد می کند که سعی می کند تفاوت بین گروه ها یا خوشه های ژنتیکی را به حداکثر برساند و در عین حال واریانس فرکانس آلل را در یک خوشه به حداقل برساند. این یک مزیت عمده در این است که هیچ فرضیات ژنتیکی جمعیت (مانند انتظارات هاردی واینبرگ) برای توصیف الگوهای تنوع ژنتیکی در بین افراد و خوشهها مورد نیاز نیست. تعاریف قبلی گروه ها برای DAPC مورد نیاز است که با استفاده از تابع find.cluster() adegenet برای پیاده سازی یک k انجام شد.-به معنای الگوریتم خوشه بندی. این تابع همچنین یک معیار اطلاعات بیزی (BIC) برای هر مقدار k ایجاد می کند که سپس برای شناسایی مقدار بهینه k استفاده می شود. در حالت ایدهآل، BIC تا رسیدن به k بهینه کاهش مییابد و سپس برای مقادیر بیشتر k افزایش مییابد . DAPC برای مقادیر k افزایش یافته از 2 به 20 با استفاده از تابع dapc() adegenet انجام شد که 75 جزء اصلی و 6 تابع متمایز را برای هر مقدار k حفظ می کند. مجموعه داده بریده شده حاوی جایگاههایی با MAF بیشتر از 0.01 نیز برای این تجزیه و تحلیل، به همان دلایلی که قبلاً توضیح داده شد، استفاده شد.
نتایج
پاکسازی مجموعه داده
در مجموع 192 SNP برای تمام 1001 نمونه ژنوتیپ شد. چهار نمونه به دلیل شکست ژنوتیپ از مجموعه داده نهایی حذف شد. هشت SNP یک شکل بودند و هفت SNP دارای ≥15٪ ژنوتیپ گم شده بودند و متعاقبا از مجموعه داده نهایی حذف شدند. در مجموع از 177 نشانگر SNP برای تجزیه و تحلیل نهایی استفاده شد، مگر اینکه خلاف آن ذکر شده باشد. 54 نشانگر SNP دارای فرکانس آللی جزئی (MAF) < 0.01 بودند.
آمار خلاصه و تنوع ژنتیکی
تنوع ژنتیکی در جمعیتهای Kudang، Walekounda (هر دو در حوضه رودخانه گامبیا)، دریاچه Sélingué (غرب حوضه نیجر) و Kongoussi (حوضه ولتا قرمز/سفید) کمترین میزان بود. جمعیت هایی که بیشترین سطح تنوع ژنتیکی را نشان دادند از مالانویل (حوضه شرقی نیجر) و دو جمعیت از منطقه جنوبی غنا (سایت VL2 از دریاچه ولتا و مخزن Afife در جنوب غنا) بودند. تعداد کل آلل ها در هر جمعیت از 257 تا 345 متغیر بود که به درصد کلی مکان های چندشکلی بر اساس جمعیت از 33.8 تا 79.7 درصد تبدیل شد. غنای آللی (R s، اندازه گیری میانگین تعداد آلل ها در هر مکان با در نظر گرفتن سوگیری های بالقوه به دلیل تفاوت در اندازه نمونه، از 1.30 تا 1.62 متغیر بود. هتروزیگوسیتی مشاهده شده بسیار شبیه به هتروزیگوسیتی مورد انتظار در همه جمعیت ها بود که در میان جایگاه ها به طور میانگین محاسبه شد (جدول 2 )، که نشان دهنده انطباق کلی با انتظارات تعادل هاردی واینبرگ (HWE) است. این توسط آزمایشات HWE توسط مکان پشتیبانی می شود، به طوری که کمتر از 5 درصد از جایگاه ها در هر جمعیت، انحراف قابل توجهی از انتظارات هاردی واینبرگ را در همه سایت ها نشان دادند ( 05/0 > P)، به استثنای ناوونی در شمال غنا (2/5 درصد مکان های نه). در HWE) و Afife Reservoir (15.1 درصد).جدول 2 آمار خلاصه تنوع ژنتیکی جمعیت های تیلاپیا نیل وحشی در سراسر آفریقای غربی ( Ho : هتروزیگوسیتی مشاهده شده ؛ H e : هتروزیگوسیتی مورد انتظار ؛ Rs : غنای آللی؛ HWE: تعادل هاردی-واینبرگ؛ N e : اندازه جمعیت موثر).جدول اندازه کامل
اندازه موثر جمعیت
اندازه جمعیت موثر ( Ne ) به طور کلی با معیارهای تنوع ژنتیکی همبستگی مثبت داشت. بیش از 50 درصد از جمعیتهای نمونهگیری شده دارای نیتروژن بیش از 100 و هیچکدام زیر 50 نبودند. یک استثنا برای این الگو، جمعیت Kongoussi در شمال بورکینافاسو بود که بیشترین N e (351.6) را نشان داد اما هتروزیگوسیتی و غنای آللی نسبتاً پایینی داشت . جدول 2 ). جزئیات کامل خلاصه و آمار تنوع ژنتیکی هر جمعیت در جدول 2 آمده است.
تمایز جمعیت
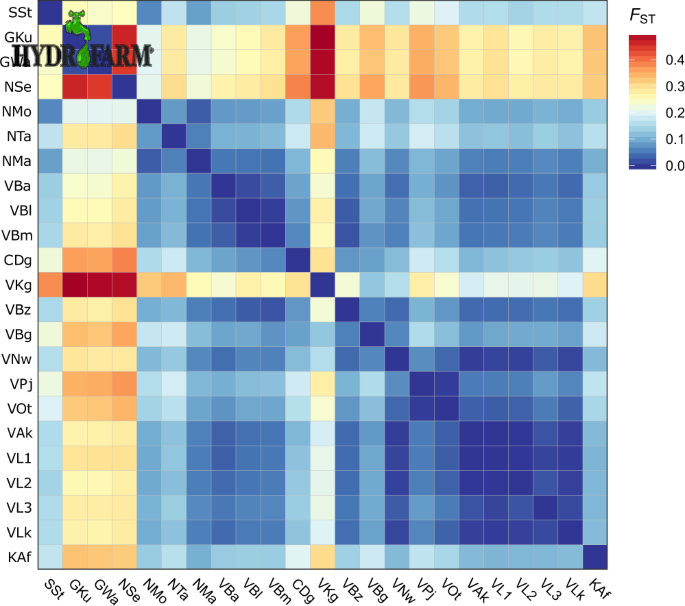
تمایز جمعیتی قابل توجهی در سطح جهانی مشاهده شد ( 001/ 0p <)، با 14.4 درصد از تنوع ژنوتیپی کلی به تفاوت بین جمعیت ها (به عنوان مثال F ST. ) زمانی که هیچ زیر گروه سلسله مراتبی به جز سطح فرد و جمعیت در نظر گرفته نشد (جدول) مشاهده شد. 3 ). هنگامی که جمعیت ها بر اساس حوضه های رودخانه با استفاده از AMOVA سلسله مراتبی گروه بندی شدند، الگوهای منطقه ای ساختار ژنتیکی بر اساس حوضه های رودخانه یا زیرحوضه ها شروع به ظهور می کنند. در برخی موارد، تفاوت بین گروه بندی منطقه ای ( F CT ) تا 13.2 درصد از تغییرات کلی را تشکیل می دهد (جدول 3 ).). گروه بندی جمعیت ها بر اساس مناطق آب و هوایی آنها می تواند تنها 1.6 درصد از تغییرات کلی را توضیح دهد. جمعیت دوتایی F ST تفاوت های قابل توجهی را در بین همه جمعیت ها نشان می دهد به استثنای چندین گروه از جمعیت های نزدیک در مناطق جنوبی دریاچه ولتا (VAk، VL1 و VL2؛ و VL3 و VLk)، دو جمعیت از رودخانه گامبیا (GKu و GWa). و دو جمعیت از رودخانه اوتی در حوضه ولتا (VOt و VPj) که پس از اصلاح بونفرونی تفاوت معنیداری با یکدیگر نشان ندادند. الگوهای واضح تمایز ژنتیکی بین جمعیتهای رودخانه گامبیا و سایر جمعیتهای نمونهگیری شده مشاهده شده است (شکل 2 )). جمعیتهای دریاچه سلینگه (غربی رودخانه نیجر) و کونگوسی (رودخانه قرمز/سفید ولتا بالایی) نیز تفاوتهای زیاد تا متوسطی را با سایر مکانها نشان دادند، با F ST جفتی از 0.2 تا 0.49 (شکل 2 ).جدول 3 تجزیه و تحلیل تغییرات مولکولی (AMOVA) ماهی تیلاپیا نیل بر اساس گروه بندی های مختلف جمعیت.جدول اندازه کامل
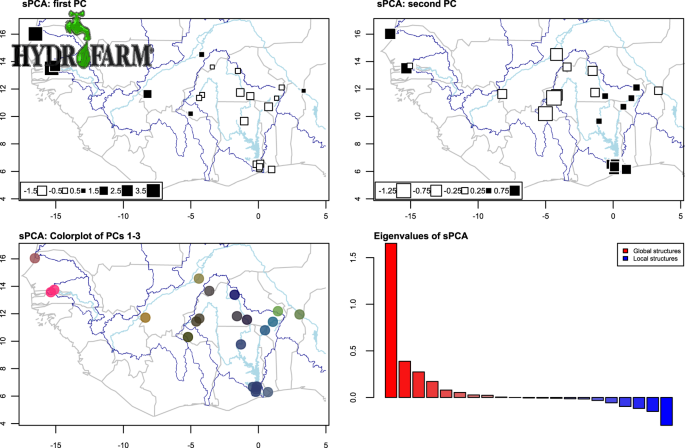
الگوهای ژنتیکی فضایی
آزمون مانتل رابطه مثبت و معنیداری را بین فواصل ژنتیکی زوجی ( F ST ) و فواصل جغرافیایی (به مواد تکمیلی مراجعه کنید) نشان داد، که نشان میدهد جداسازی فاصله به فاصله تأثیر متوسطی بر تمایز ژنتیکی در مقیاس منطقهای دارد ( r = 0.521، p = 0.001 ). ). مطابق با این، sPCA ساختار فضایی جهانی قابل توجهی ( p = 0.03) و ساختار محلی غیر قابل توجه ( p = 0.97) را به طور کلی نشان داد، که نشان می دهد شباهت ژنتیکی جمعیت ها با نزدیکی جغرافیایی آنها به یکدیگر همبستگی مثبت دارد. با این حال، در حوضه ولتا، یک رابطه قابل توجه و در عین حال بسیار ضعیفتر بین فواصل ژنتیکی و جغرافیایی وجود داشت ( r = 0.196، p = 0.029)، نشان می دهد که جداسازی بر اساس فاصله یک عامل نسبتاً جزئی است که تمایز ژنتیکی را در بین جمعیت ها شکل می دهد. الگوهای تفاوتها و شباهتهای ژنتیکی در سراسر آفریقای غربی زمانی آشکار میشوند که نمرات عقبافتاده سه جزء اصلی اولیه sPCA به صورت فضایی ترسیم شوند (شکل 3 ). یک “نقاط رنگی” که نشان دهنده نمرات عقب افتاده سه جزء اصلی هر جمعیت در فضای رنگی قرمز-سبز-آبی است، شباهت ژنتیکی را در جمعیت های حوضه رودخانه نیجر، جمعیت رودخانه اوتی و دریاچه ولتا، قرمز/ برجسته می کند. جمعیت رودخانه ولتا سفید و ولتا سیاه و جمعیت رودخانه گامبیا (شکل 3 ).
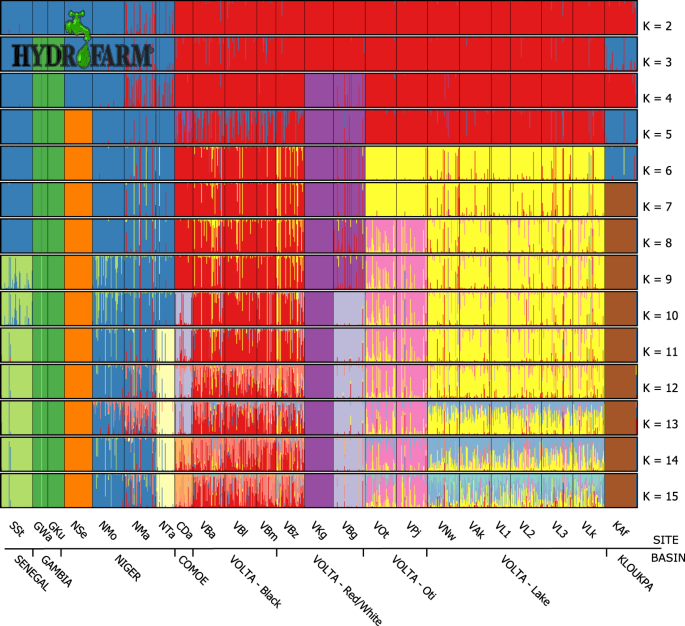
خوشه بندی ژنتیکی فردی
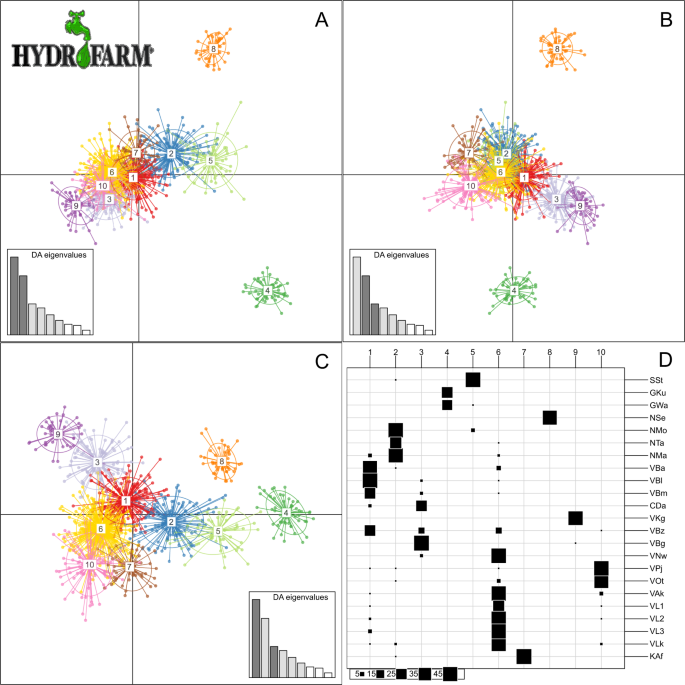
رویکردهای مبتنی بر بیزی (ساختار) و چند متغیره (DAPC) الگوهای بسیار سازگار خوشهبندی ژنتیکی فردی را برای مقادیر مختلف k از 2 تا 15 نشان دادند. برای وضوح، فقط نتایج DAPC نشان داده شدهاند. نمودارهای احتمالات خلفی فردی برای انتساب به یک خوشه مشخص نشان می دهد که الگوهای تمایز ژنتیکی بر اساس حوضه رودخانه در مقادیر مختلف k آشکار است (شکل 4 ). محتملترین تعداد خوشهها ( k ) برای رویکرد بیزی، بر اساس روش دلتا K 33 ، چهار بود، اگرچه یک پیک ثانویه در دلتا K نیز برای k = 13 دیده میشود که میتواند ساختار ژنتیکی در مقیاس دقیقتر را نشان دهد. تغییرات در BIC نشان داد که یک مقدار بهینه ازk برای استفاده برای DAPC احتمالاً بین 10 و 15 خواهد بود. تغییرات جزئی در BIC و Delta K و مقایسه بین نتایج DAPC و SRUCTURE را می توان در مواد تکمیلی یافت. خوشهبندی در پایینترین سطح ( k = 2) تمایزی را بین جمعیتهای حوضه ولتا و سایر جمعیتها نشان میدهد، به ویژه برای تجزیه و تحلیل DAPC (شکل 4 ). به عنوان تعداد خوشه ها ( k) افزایش مییابد، خوشهبندی فردی با توجه به حوضه رودخانه از نزدیک دنبال میشود. در حوضه ولتا، چهار خوشه ژنتیکی را می توان مشاهده کرد و می توان آن را به عنوان یک خوشه دریاچه ولتا (شامل ناوونی، که روی رودخانه ولتا سفید قرار دارد)، یک خوشه رودخانه اوتی، یک خوشه ولتا سیاه و یک خوشه ولتا قرمز/سفید توصیف کرد. تفاوت های نسبی بین خوشه های شناسایی شده با DAPC برای k = 10 در شکل 5 نشان داده شده است . این نشان میدهد که بزرگترین تفاوتهای ژنتیکی در بین همه خوشهها از دو خوشه متشکل از افرادی است که از دریاچه سلینگه (رودخانه نیجر) و از دو جمعیت رودخانه گامبیا هستند.
بحث
تجزیه و تحلیل های متعدد انجام شده در این مطالعه همگی الگوی ساختار ژنتیکی فضایی و تمایز جمعیت ماهی تیلاپیا نیل در سراسر آفریقای غربی را تایید می کند. الگوهای جداسازی در مقیاس وسیع با همبستگی فضایی قابل توجه فرکانسهای آللی جمعیت شناساییشده توسط sPCA سازگار است. تمایز ژنتیکی O. niloticusدر سراسر آفریقای غربی به طور کلی می توان با حوضه های رودخانه، و تا حدی، زیرحوضه ها، با جریان ژن در میان جمعیت ها به دنبال الگوهای جداسازی فاصله توضیح داد. با این حال، شباهت ژنتیکی در جمعیتهایی که بیش از 1400 کیلومتر از هم به هم متصل شدهاند توسط رودخانه نیجر (مثلاً Malanville و Mopti) شواهدی را ارائه میدهد که جریان ژنی به اندازه کافی در امتداد یک آبراه بزرگ و پیوسته میتواند در بازههای زمانی مرتبط ژنتیکی رخ دهد. در مقابل، در فاصله بسیار کوتاهتری در اطراف مناطق رودخانه قرمز و سفید ولتا، ناهمگونی ژنتیکی را در میان چندین جمعیت (بازگا، دریاچه باگره و ناوونی) مشاهده میکنیم، که نشان میدهد تفاوتهای جمعیتی میتواند در جغرافیای نسبتاً نزدیک باقی بماند.
مطالعه ما پیچیدگی جمعیت اضافی را نشان می دهد که قبلاً مستند نشده است. واگراترین جمعیت ها در میان نمونه برداری ها از دریاچه سلینگه در حوضه نیجر و از رودخانه گامبیا بود که تمایز شدیدی را با سایر جمعیت ها نشان داد. بر خلاف Rognon & Guyomard 8 ، که مشاهده کردند که جمعیت O. niloticus از سنگال بیشتر از سایر جمعیت های غرب آفریقا متفاوت است، ما یک شباهت ژنتیکی بین جمعیت های سنگال و جمعیت های رودخانه بالایی و میانی نیجر مشاهده می کنیم. این با این باور مطابقت دارد که پس از آخرین بارندگی، در حدود 12000 تا 7500 سال پیش، رودخانه های سنگال و گامبیا توسط جانوران آب شیرین از حوضه نیجر 38 مجدداً مستعمره شدند.. به طور مشابه، رودخانههای بلک ولتا و پنجاری احتمالاً در گذشته 38 از شاخههای رودخانه نیجر بودهاند ، که ممکن است تمایز ژنتیکی متوسط مشاهدهشده در میان محلهای نمونهبرداری که سه شاخه اصلی حوضه ولتا را پوشش میدهند توضیح دهد.
ما تنوع ژنتیکی با استفاده از SNP را در اکثر جمعیتهای نمونهگیری شده مشابه مییابیم، به استثنای چندین جمعیت که تنوع ژنتیکی کمتری نسبت به سایرین نشان میدهند. اکثر جمعیتها هتروزیگوسیتی SNP بیشتری نسبت به آنچه قبلاً برای حوضه ولتا 18 و در دو جمعیت تیلاپیا پرورش یافته انتخابی گزارش شده بود نشان دادند ، البته با استفاده از آرایههای SNP که لزوماً برای اطلاعرسانی در غرب آفریقا بهینه نشدهاند. تنوع ژنتیکی کمتر در رودخانه گامبیا و منطقه غربی دورتر رودخانه نیجر با الگوهایی که اغلب در محدوده گونهای یا جمعیتهای جدا شده دیده میشوند، سازگار است. Rognon & Gouyomard 8کاهش تنوع آنزیمی را در نمونههای تیلاپیا نیل از رودخانه سنگال یافت و اثرات پایهگذار در حاشیه توزیع را به عنوان توضیح احتمالی ذکر کرد. اثرات بنیانگذار ممکن است الگوی تمایز ژنتیکی و کاهش تنوع شناسایی شده در رودخانه گامبیا را توضیح دهد. با این حال، در مقابل، ما سطوح نسبتاً بالایی از تنوع SNP را در جمعیت رودخانه سنگال یافتیم، که ممکن است نشان دهد این منطقه از جمعیت های بزرگتر و پایدارتر در مقایسه با رودخانه گامبیا در نزدیکی آن پشتیبانی می کند. این با تفاوت در Ne تأیید می شود ، که در غیاب جریان ژن منجر به نرخ های متفاوت از دست دادن تنوع از طریق رانش ژنتیکی می شود. از طرف دیگر، رودخانه سنگال ممکن است ارتباط تاریخی متناوبتری با حوضه نیجر داشته باشد .، احتمالاً تأثیرات رانش ژنتیکی بر کاهش تنوع را تعدیل می کند. دو جمعیت در دریاچه Sélingué در رودخانه نیجر بالایی، و Kongoussi در رودخانه Red Volta بالایی نیز درصد کمتری از مکانهای چندشکلی را نسبت به سایرین نشان میدهند. این ممکن است در نتیجه موقعیت جغرافیایی آنها در قسمت بالایی سیستم های رودخانه مربوطه آنها باشد که باعث انزوای نسبی می شود. در نتیجه، محدودیت جریان ژن بین سایر جمعیت ها یا افزایش قرار گرفتن در معرض نوسانات فصلی و متناوب در جریان آب می تواند افزایش احتمالی نرخ از دست دادن تنوع ژنتیکی ناشی از رانش ژنتیکی را توضیح دهد.
گسترش سریع جمعیت در نتیجه توده های آبی جدید ایجاد شده توسط سدها به طور بالقوه می تواند تأثیر قابل توجهی بر ویژگی های ژنتیکی یک جمعیت داشته باشد 41 . دریاچه ولتا که پس از تکمیل سد آکوسومبو در سال 1965 ایجاد شد، بزرگترین دریاچه ساخته دست بشر در جهان از نظر مساحت است و 8502 کیلومتر مربع وسعت دارد .. علیرغم چنین مناطق وسیعی و احتمال گسترش عظیم جمعیت تیلاپیا نیل در سرتاسر دریاچه پس از تشکیل آن، ما هیچ شواهدی مبنی بر خروج از تعادل هاردی واینبرگ (HWE) و تمایز ژنتیکی کمی در میان جمعیتهای دریاچه ولتا، با فاصله بیش از 400 کیلومتری دریاچه، کشف نکردیم. این می تواند به این معنی باشد که یا در نتیجه تشکیل دریاچه ولتا، اثر بنیانگذار محدودی بر روی جمعیت تیلاپیا نیل وجود داشته است و ساختار ژنتیکی این جمعیت ها تغییر نکرده است. روش دیگر، هر گونه تمایز ژنتیکی قبل یا پس از ساخت سد آکوسومبو با جریان ژنی کافی در سراسر دریاچه یکسان شده است و به تعادل فراوانی آللی رسیده است. در تمام سایتها، تنها جمعیتی از مخزن Afife خروج HWE قابل توجهی را نشان دادند.1 . انحراف از HWE همراه با تمایز ژنتیکی متوسط و اندازه جمعیت موثر نسبتا کم که در مخزن Afife مشاهده میشود، نشانهای از اثرات بنیانگذار است که به تازگی ایجاد شده است، احتمالاً از جمعیتهای رودخانه ولتا در نزدیکی.
مطالعات ژنتیکی جمعیتی قبلی ماهی تیلاپیا نیل، گسستهای عمدهای را در توزیع طبیعی آن شناسایی کرده است که سه گروه کلان جغرافیایی را مشخص میکند: (1) جمعیتهای سودانو-ساحلی، پوشش غرب آفریقا، (ب) جمعیتهای دره ریفت اتیوپی و (iii) زهکشی نیل و کنیا. جمعیت دره ریفت 4 ، 5 . مطالعه ما نسبت به برآوردهای قبلی بر اساس نشانگرهای ریزماهواره (R ST = 0.09) در میان جمعیتهای منطقه سودانو-ساحلی، F ST کلی بیشتری را شناسایی میکند.. این میتواند به دلیل پوشش نمونهبرداری بیشتر باشد، بهویژه گنجاندن جمعیتهایی از رودخانه گامبیا، که تفاوت زیادی با سایر جمعیتهای نمونهگیری شده دارند. با این حال، این ممکن است به دلیل تفاوت در هتروزیگوسیتی مورد انتظار ناشی از نشانگرهای ریزماهواره بسیار چندشکلی در مقایسه با نشانگرهای SNP دو شکل باشد، که میتواند بر کرانهای بالایی مقادیر احتمالی F ST که میتوان به دست آورد تأثیر بگذارد . مطالعه ما اولین تخمین تنوع جمعیت را بر اساس نشانگرهای SNP در غرب آفریقا ارائه میکند، که یک خط پایه مهم را ارائه میکند زیرا SNP به طور فزاینده نشانگر انتخابی برای مطالعات جمعیت و برنامههای پرورش آبزی پروری تجاری است.
نوسانات آب و هوایی در منطقه ساحل می تواند در طول سال چشمگیر باشد، به ویژه در مقایسه با مناطق گرمسیری جنوب غنا، که ممکن است فشار انتخابی بیشتری را برای صفات تجاری مهم مانند تحمل دما و شوری اعمال کند. محدوده دما از 12 تا 47 درجه سانتیگراد در طول سال در بورکینافاسو 43 ، در تضاد با دمایی است که به ندرت از 21 تا 34 درجه سانتیگراد در جنوب غنا فراتر می رود ، که دامنه های حرارتی بسیار متفاوت را در محدوده طبیعی O. niloticus برجسته می کند.. اگرچه AMOVA سلسله مراتبی بر اساس گروهبندی جمعیتها بر اساس منطقه آب و هوایی آنها، تنوع زیادی را به اندازه گروهبندیهای مبتنی بر حوضههای رودخانه توضیح نمیدهد، وجود گروههایی از جمعیتهای جدا شده ژنتیکی در سراسر منطقه ساحل شناساییشده در این مطالعه، مانند دریاچه Sélingué و Kongoussi، در این زمینه از علاقه خاصی برخوردار هستند. شایان ذکر است، جمعیت کنگوسی احتمالاً به دلیل تبادل کم آب، دورههای طولانی بارندگی صفر و کمعمق بودن آن در سراسر آن منطقه، نوسانهایی در شرایط محیطی بهطور قابلتوجهی بیشتر از جمعیت رودخانههای اصلی تجربه میکند. اخیراً جمعیتهای منحصربهفرد ژنتیکی که منابع ژنتیکی بالقوه ارزشمندی هستند در سایر مناطق پراکنش طبیعی ماهی تیلاپیا نیل، به ویژه در شرق آفریقا کشف شده است .. نتایجی مانند این مبنایی برای تحقیقات عمیق تر در مورد اینکه آیا جمعیت های ژنتیکی متمایز منابع ژنتیکی بالقوه را برای استفاده در برنامه های اصلاحی تجاری در آینده نشان می دهند، فراهم می کند. مطالعات ژنتیکی آینده برای هدف قرار دادن شدت نمونهبرداری بیشتر و پوشش جغرافیایی در مقیاس دقیقتر در اطراف مناطقی که جمعیتهای ژنتیکی متمایز در آن حضور دارند، مناسب خواهد بود. مکمل این، تحقیقاتی که بر درک اینکه آیا چنین تمایزی منجر به هر گونه تفاوت فیزیولوژیکی در سطح جمعیت می شود، بسیار ارزشمند خواهد بود.
در پرداختن به سوالات تنوع ژنتیکی و ساختار ژنتیکی جمعیت تیلاپیا نیل غرب آفریقا، پیامدهای بالقوه بر صنعت رو به رشد پرورش ماهی تیلاپیا در سراسر منطقه باید در نظر گرفته شود. ما نشان میدهیم که سطوح بالایی از تنوع ژنتیکی در میان جمعیتهای واقع در اکثر حوضههای آبی اصلی وجود دارد، و برخی از جمعیتها را شناسایی میکنیم که از سایرین بسیار متمایز هستند، که نشاندهنده حضور جمعیتهای منحصربهفرد ژنتیکی است. نتایج ما نشان میدهد که تنوع ژنتیکی فراوانی در سراسر جمعیتهای تیلاپیا نیل وحشی در غرب آفریقا برای حمایت از ایجاد و توسعه بلندمدت برنامههای اصلاحی انتخابی برای آبزی پروری در منطقه وجود دارد. با این حال، پیامد یک صنعت بزرگ آبزی پروری مبتنی بر جمعیت های اهلی شده و انتخابی، این است که استفاده و توزیع گسترده این حیوانات اجتناب ناپذیر است. این ممکن است تأثیرات بالقوهای بر جمعیتهای طبیعی داشته باشد که حیوانات پرورشی به طور ناخواسته فرار کنند و در سایر بخشهای آفریقا موضوعی رو به رشد است.17 ، 45 ، 46 ، 47 ، 48 . دانش ژنتیک جمعیت محلی که از طریق مطالعاتی مانند این (احتمالاً با استفاده از ابزارهای هدفمندتر ژنوتیپ SNP) ایجاد شده است، نقش مهمی در شناسایی، تجزیه و تحلیل و کمک به مدیریت اثرات احتمالی فرارهای احتمالی دارد. کار اخیر در شناسایی نشانگرهای SNP گونهای خاص در میان گونههای تیلاپیا نزدیک به هم میتواند در جاهایی که همپوشانیهای جغرافیایی اتفاق میافتد، و پتانسیل هیبریداسیون وجود دارد .
نتیجه گیری
این مطالعه ساختار و تمایز ژنتیکی واضحی را در میان جمعیتهای تیلاپیا نیل غرب آفریقا شناسایی میکند، که با الگوهای وسیعی که در مطالعات قبلی یافت شدهاند مطابقت دارد ( 4 ، 5 ، 8 ) . علاوه بر این، این مطالعه شواهد جدیدی برای ساختار ژنتیکی در مقیاس ریز در حوضه ولتا و برای جمعیتهای متفاوت ژنتیکی بیشتر در گامبیا ارائه میکند. الگوهای ژنتیکی فضایی و روشهای خوشهبندی فردی نشان میدهد که ساختار ژنتیکی را میتوان تا حد زیادی توسط رودخانهها و زیرحوضههای اصلی در سراسر منطقه توضیح داد. مجموعه نشانگر 192 SNP مورد استفاده در این مطالعه یک ابزار موثر برای افتراق جمعیت ماهی تیلاپیا است و توصیه می کنیم این مجموعه نشانگر را در غربالگری جمعیت آتی O.niloticus بگنجانید.، به طور بالقوه در ارتباط با سایر SNP های خاص گونه. نتایج ما اساس یک پلت فرم محکم برای تحقیقات آینده در مورد منابع ژنتیکی تیلاپیا وحشی در غرب آفریقا و شناسایی ژرم پلاسم بالقوه ارزشمند برای استفاده در برنامههای پرورش آبزی پروری را تشکیل میدهد.
در دسترس بودن داده ها
مجموعه داده و فراداده کامل این نشریه در مخزن دیجیتال دریاد ( https://doi.org/10.5061/dryad.bk3j9kd6m ) در دسترس است.
منابع
- Trewevas، E. Tilapiine ماهیان از جنس Sarotherodon، Oreochromis و Danakilia . 583 (موزه بریتانیا (تاریخ طبیعی)، 1983).
- Rognon، X.، Andriamanga، M.، McAndrew، B. & Guyomard، R. تنوع آلوزیم در جمعیت های طبیعی و کشت شده در دو گونه ماهی تیلاپیا: Oreochromis niloticus و Tilapia zillii . وراثت 76 ، 640-650 (1996).مورد مقاله Google Scholar
- Rognon، X. و Guyomard، R. تمایز DNA میتوکندری در میان جمعیت تیلاپیا نیل شرق و غرب آفریقا. مجله زیست شناسی ماهی 51 ، 204-207 (1997).مورد مقاله Google Scholar
- Agnese، J.-F.، Adepo-Gourene، B.، Abban، EK & Fermon، Y. تمایز ژنتیکی در میان جمعیت های طبیعی ماهی تیلاپیا نیل Oreochromis niloticus (Teleostei، Cichlidae). وراثت 79 ، 88-96 (1997).مقاله Google Scholar
- Bezault، E. و همکاران . تنوع مکانی و زمانی در ساختار ژنتیکی جمعیت تیلاپیا وحشی نیل ( Oreochromis niloticus ) در سراسر آفریقا. BMC Genetics 12 ، 102 (2011).مقاله Google Scholar
- Eknath، AE & Hulata، G. استفاده و تبادل منابع ژنتیکی ماهی تیلاپیا نیل ( Oreochromis niloticus ). بررسی در آبزی پروری 1 ، 197-213 (2009).مقاله Google Scholar
- Nyingi، D.، De Vos، L.، Aman، R. & Agnèse، J.-F. خصوصیات ژنتیکی یک جمعیت بومی ناشناخته و در خطر انقراض نیل tilapia Oreochromis niloticus (Linnaeus، 1758) (Cichlidae؛ Teleostei) در مرداب Loboi (کنیا). آبزی پروری 297 ، 57-63 (2009).مورد مقاله Google Scholar
- Rognon, X. & Guyomard, R. میزان زیادی از انتقال DNA میتوکندری از Oreochromis aureus به O. niloticus در غرب آفریقا. اکولوژی مولکولی 12 ، 435-445 (2003).مورد مقاله Google Scholar
- Nyingi، DW & Agnese، J.-F. هیبریداسیون داخلی اخیر با انتقال mtDNA انحصاری از Oreochromis leucostictus (Trewavas، 1933) به Oreochromis niloticus (Linnaeus، 1758) در دریاچه Baringo، کنیا آشکار شد. مجله زیست شناسی ماهی 70 ، 148-154 (2007).مورد مقاله Google Scholar
- فائو سالنامه فائو – آمار شیلات و آبزی پروری 2017 . (سازمان خواربار و کشاورزی ملل متحد، 2019).
- ایکنات، AE و همکاران . بهبود ژنتیکی تیلاپیاهای پرورشی: عملکرد رشد هشت سویه Oreochromis niloticus آزمایش شده در محیط های مختلف مزرعه. آبزی پروری 111 ، 171-188 (1993).مقاله Google Scholar
- Dey, MM تأثیر تیلاپیا نیل اصلاح شده ژنتیکی در آسیا. اقتصاد و مدیریت آبزی پروری 4 ، 107–124، https://doi.org/10.1080/13657300009380263 (2000).مقاله Google Scholar
- بانک توسعه آسیایی ارزیابی تأثیر توسعه تیلاپیا پرورشی اصلاح شده ژنتیکی و انتشار آنها در کشورهای منتخب . (بانک توسعه آسیایی، 2005).
- Attipoe، FYK Breeding و انتخاب برای سویههای رشد سریعتر ماهی تیلاپیا نیل، Oreochromis niloticus، در غنا ، پایاننامه دکترا، دانشگاه کیپ کوست، غنا (2006).
- Attipoe، FYK & Abban، EK در تنوع زیستی، مدیریت و استفاده از ماهیان غرب آفریقا (ویرایشگران Eddie K. Abban، Christine Marie V. Casal، Patrick Dugan و Thomas M. Falk) 16-17 (مجموعه مقالات کنفرانس مرکز جهانی ماهی، 2004) .
- Ponzoni، RW و Brummett، RE در مدیریت منابع ژنتیکی ماهی و برنامه های انتشار بذر برای آفریقا، مقاله گاه به گاه CIFAA شماره 29 (ویرایش های Devin M. Bartley و همکاران ) 32-41 (FAO، 2008).
- Lind، CE، Brummett، RE & Ponzoni، RW بهره برداری و حفاظت از منابع ژنتیکی ماهی در آفریقا: مسائل و اولویت ها برای توسعه و تحقیق آبزی پروری. بررسی در آبزی پروری 4 ، 125-141 (2012).مقاله Google Scholar
- Van Bers، NEM، Crooijmans، RPMA، Groenen، MAM، Dibbits، BW & Komen، J. SNP تشخیص و تعیین ژنوتیپ در ماهی تیلاپیا. منابع بوم شناسی مولکولی 12 ، 932-941، https://doi.org/10.1111/j.1755-0998.2012.03144.x (2012).مقاله PubMed Google Scholar
- جوشی، آر و همکاران . توسعه و اعتبار سنجی 58K SNP-Array و نقشه پیوند با چگالی بالا در نیل Tilapia ( O. niloticus ). مرزها در ژنتیک 9 ، https://doi.org/10.3389/fgene.2018.00472 (2018).
- Conte، MA، Gammerdinger، WJ، Bartie، KL، Penman، DJ & Kocher، TD مجموعه ای با کیفیت بالا از ژنوم تیلاپیا نیل ( Oreochromis niloticus ) ساختار دو ناحیه تعیین جنسیت را نشان می دهد. BMC Genomics 18 ، 341، https://doi.org/10.1186/s12864-017-3723-5 (2017).مورد مقاله PubMed PubMed Central Google Scholar
- گیون، آر و همکاران . نقشه ای با وضوح بالا از ژنوم تیلاپیا نیل: منبعی برای مطالعه سیکلیدها و سایر پرکومورف ها. BMC Genomics 13 , 222 (2012).مورد مقاله Google Scholar
- Jombart، T. adegenet: یک بسته R برای تجزیه و تحلیل چند متغیره نشانگرهای ژنتیکی. Bioinformatics 24 ، 1403–1405، https://doi.org/10.1093/bioinformatics/btn129 (2008).مورد مقاله PubMed PubMed Central Google Scholar
- Goudet، J. hierfstat، بسته ای برای r برای محاسبه و آزمایش آمارهای F سلسله مراتبی. یادداشتهای اکولوژی مولکولی 5 ، 184-186، https://doi.org/10.1111/j.1471-8286.2004.00828.x (2005).مقاله Google Scholar
- Benjamini, Y. & Hochberg, Y. کنترل نرخ کشف نادرست: یک رویکرد عملی و قدرتمند برای آزمایش چندگانه. مجله انجمن آماری سلطنتی (سری B) 57 ، 289-300 (1995).MathSciNet ریاضی Google Scholar
- تیم اصلی R. (بنیاد R برای محاسبات آماری، وین، اتریش، 2013).
- Waples، RS یک تصحیح سوگیری برای تخمین اندازه جمعیت مؤثر بر اساس عدم تعادل پیوندی در مکانهای ژنی غیر مرتبط. Conservation Genetics 7 ، 167-184، https://doi.org/10.1007/s10592-005-9100-y (2006).مقاله Google Scholar
- Do, C. et al . NeEstimator v2: اجرای مجدد نرم افزار برای تخمین اندازه جمعیت موثر معاصر (Ne) از داده های ژنتیکی. منابع اکولوژی مولکولی 14 ، 209-214، https://doi.org/10.1111/1755-0998.12157 (2014).مورد مقاله PubMed Google Scholar
- Excoffier، L. & Lischer، مجموعه HEL Arlequin نسخه 3.5: سری جدیدی از برنامه ها برای انجام تجزیه و تحلیل ژنتیک جمعیت تحت لینوکس و ویندوز. منابع اکولوژی مولکولی 10 ، 564–567 https://doi.org/10.1111/j.1755-0998.2010.02847.x .مقاله Google Scholar
- فسیل Vavrek، MJ : ابزارهای تجزیه و تحلیل دیرینه اکولوژیکی و جغرافیایی دیرینه. Palaeontologia Electronica ، 14.11.11T (2011).
- Jombart, T., Devillard, S., Dufour, AB & Pontier, D. آشکارسازی الگوهای فضایی مرموز در تنوع ژنتیکی با یک روش چند متغیره جدید. وراثت 101 ، 92-103 (2008).مورد مقاله Google Scholar
- Pritchard, JK, Stephens, M. & Donnelly, P. Inference of Population Structure Using Multilocus Genotype Data. ژنتیک 155 ، 945-959 (2000).مورد PubMed PubMed Central Google Scholar
- فالوش، دی.، استفنز، ام و پریچارد، JK استنتاج ساختار جمعیت با استفاده از دادههای ژنوتیپ چند لوکوس: مکانهای پیوندی و فرکانسهای آللی همبسته. ژنتیک 164 ، 1567-1587 (2003).مورد PubMed PubMed Central Google Scholar
- Evanno, G., Regnaut, S. & Goudet, J. تشخیص تعداد خوشههای افراد با استفاده از نرمافزار SRUCTURE: یک مطالعه شبیهسازی. اکولوژی مولکولی 14 ، 2611-2620، https://doi.org/10.1111/j.1365-294X.2005.02553.x (2005).مورد مقاله Google Scholar
- Earl, DA & vonHoldt, BM STRUCTURE HARVESTER: وب سایت و برنامه ای برای تجسم خروجی ساختار و پیاده سازی روش Evanno. منابع ژنتیک حفاظت 4 ، 359–361، https://doi.org/10.1007/s12686-011-9548-7 (2012).مقاله Google Scholar
- Jakobsson، M. & Rosenberg، NA CLUMPP: یک برنامه تطبیق و جایگشت خوشه برای مقابله با تغییر برچسب و چندوجهی در تجزیه و تحلیل ساختار جمعیت. Bioinformatics 23 ، 1801–1806، https://doi.org/10.1093/bioinformatics/btm233 (2007).مورد مقاله PubMed PubMed Central Google Scholar
- روزنبرگ، NA distruct: برنامه ای برای نمایش گرافیکی ساختار جمعیت. یادداشتهای اکولوژی مولکولی 4 ، 137-138، https://doi.org/10.1046/j.1471-8286.2003.00566.x (2004).مقاله Google Scholar
- Jombart, T., Devillard, S. & Balloux, F. تجزیه و تحلیل متمایز اجزای اصلی: روشی جدید برای تجزیه و تحلیل جمعیت های ساختار یافته ژنتیکی. BMC Genetics 11 ، 94 (2010).مقاله Google Scholar
- Léveque، C. پویایی و حفاظت از تنوع زیستی: ماهی های آب شیرین آفریقای استوایی . 402 (انتشارات دانشگاه کمبریج، 1997).
- Lind، CE، Kilian، A. & Benzie، JAH توسعه نشانگرهای فناوری آرایههای تنوع به عنوان ابزاری برای ارزیابی سریع ژنومی در تیلاپیا نیل، Oreochromis niloticus . Animal Genetics 48 ، 362-364، https://doi.org/10.1111/age.12536 (2017).مورد مقاله PubMed Google Scholar
- Thieme، ML و همکاران . اکومناطق آب شیرین آفریقا و ماداگاسکار: ارزیابی حفاظت . (آیلند پرس، واشنگتن دی سی، 2005).
- Neraas، LP & Spruell، P. تکه تکه شدن سیستم های رودخانه ای: اثرات ژنتیکی سدها بر قزل آلای گاو نر ( Salvelinus confluentus ) در سیستم رودخانه کلارک فورک. اکولوژی مولکولی 10 ، 1153-1164، https://doi.org/10.1046/j.1365-294X.2001.01269.x (2001).مورد مقاله PubMed Google Scholar
- Jakobsson, M., Edge, MD & Rosenberg, NA رابطه بین FST و فرکانس آلل فراوان. ژنتیک 193 ، 515-528، https://doi.org/10.1534/genetics.112.144758 .مقاله Google Scholar
- نسخههای JA Jeune Afrique Atlases: Atlas بورکینافاسو . ویرایش سوم، (Les Editions JA، 1998).
- WeatherSpark. میانگین آب و هوا برای آکرا، غنا ، https://weatherspark.com/averages/28541/Accra-Greater-Accra-Ghana (2014).
- Ansah, YB, Frimpong, EA & Hallerman, EM سویه های تیلاپیا ژنتیکی بهبود یافته در آفریقا: مزایای بالقوه و اثرات منفی. پایداری 6 ، 3697-3721 (2014).مقاله Google Scholar
- Deines، AM، Bbole، I.، Katongo، C.، Feder، JL & Lodge، DM هیبریداسیون بین گونه های بومی Oreochromis و تیلاپیا نیل O. niloticus در رودخانه کافو، زامبیا معرفی شده است. آفریقایی . مجله علوم آبزیان 39 ، 23–34، https://doi.org/10.2989/16085914.2013.864965 (2014).مقاله Google Scholar
- Firmat, C., Alibert, P., Losseau, M., Baroiller, J.-F. & Schliewen، هیبریداسیون بین گونه ای و ساختار جمعیت با واسطه تهاجم متوالی در سیچلاید Oreochromis mossambicus در حال انقراض. PLOS ONE 8 ، e63880، https://doi.org/10.1371/journal.pone.0063880 (2013).تبلیغات مورد مقاله PubMed PubMed Central Google Scholar
- Shechonge، A. و همکاران . استعمار گسترده حوضه های آبریز تانزانیا توسط ماهی های تیلاپیا Oreochromis معرفی شده: میراث ده ها سال معرفی عمدی. Hydrobiologia ، https://doi.org/10.1007/s10750-018-3597-9 (2018).مقاله Google Scholar
- سیایفودین، ام. و همکاران . کشف نشانگر خاص گونه در تیلاپیا. گزارش های علمی 9 ، 13001، https://doi.org/10.1038/s41598-019-48339-2 (2019).تبلیغات مورد مقاله PubMed PubMed Central Google Scholar
سپاسگزاریها
نویسندگان مایلند از افراد زیر برای مشارکت ارزشمندشان در جمع آوری نمونه تشکر و قدردانی کنند: از بنین: Arsène d’Almeida، توگو: M. Agbokoussé; بورکینافاسو: Bema Ouattara; مالی: M. Sanogo; غنا: اولگا آسمیان، لیلی کونادو؛ ساحل عاج: دکتر ارنست کواکو، اولگا آسمیان. سنگال: M. Manel; گامبیا: Moustapha Kébé، Musasaihou Mbenga، Njie Mariatou، Fatou Drammeh. ما همچنین از جان بنزی و دوین بارتلی برای نظرات مفید در مورد نسخه قبلی نسخه خطی تشکر می کنیم. این مطالعه با کمک مالی آژانس اسپانیایی همکاری بینالمللی/فائو TIVO پروژه GCP/RAF/417/SPA، از کمیسیون اروپا (گرنت شماره 2000000466) به عنوان کمک به برنامه تحقیقاتی CGIAR در مورد دام و ماهی، و از طرف دفتر زیر منطقه ای فائو برای غرب آفریقا (SFW).
درباره ی نویسنده
وابستگی ها
- WorldFish، Jalan Batu Maung، Batu Maung، 11960، Bayan Lepas، Penang، مالزیکرتیس ای لیند
- CSIRO کشاورزی و غذا، Castray Esplanade، هوبارت، استرالیاکرتیس ای لیند
- مرکز تحقیقات و توسعه آبزی پروری (ARDEC)، موسسه تحقیقات آب CSIR، صندوق پستی 139، آکوسومبو، غناSeth K. Agyakwah & Felix Y. Attipoe
- سازمان خواربار و کشاورزی ملل متحد (فائو)، رم، ایتالیاکریستوفر نوجنت
- دانشگاه و تحقیقات واگنینگن، اصلاح نژاد و ژنومیک حیوانات، واگنینگن، هلندریچارد پی ام ای کروییمنز
- مرکز بینالمللی تحقیق و توسعه در مورد دام در منطقه نیمه مرطوب (CIRDES) 01 BP 454 Bobo-Dioulasso 01, Bobo-Dioulasso, بورکینافاسوابوباکار توگوینی
- دانشگاه نازی BONI (UNB) 01 BP 1091 Bobo-Dioulasso 01, Bobo-Dioulasso, بورکینافاسوابوباکار توگوینی
مشارکت ها
CL طراحی مطالعه را رهبری کرد، تجزیه و تحلیل داده ها را انجام داد و پیش نویس دستنوشته را نوشت. AT در طراحی مطالعه، جمعآوری نمونه شرکت کرد و بر استخراج DNA نظارت داشت. SA و FA در طرح مطالعه و مجموعه نمونه شرکت کردند. CN در طراحی مطالعه شرکت کرد، هماهنگی کرد و در جمع آوری نمونه شرکت کرد. RC سنجش SNP را طراحی کرد و بر فعالیتهای ژنوتیپسازی نظارت کرد. همه نویسندگان مقاله را بررسی کردند.
نویسنده متناظر
اعلامیه های اخلاقی
منافع رقابتی
نویسندگان اظهار کردند که هیچ علاقه ای به رقابت ندارند.
اطلاعات تکمیلی
یادداشت ناشر Springer Nature با توجه به ادعاهای قضایی در نقشه های منتشر شده و وابستگی های سازمانی بی طرف باقی می ماند.
اطلاعات تکمیلی
مجموعه داده 1
اطلاعات تکمیلی 2
اطلاعات تکمیلی 3
حقوق و مجوزها
دسترسی آزاد این مقاله دارای مجوز Creative Commons Attribution 4.0 بینالمللی است که اجازه استفاده، اشتراکگذاری، اقتباس، توزیع و تکثیر را در هر رسانه یا قالبی میدهد، تا زمانی که اعتبار مناسبی را به نویسنده (نویسندگان) اصلی و منبع بدهید. پیوندی به مجوز Creative Commons ارائه دهید و نشان دهید که آیا تغییرات ایجاد شده است یا خیر. تصاویر یا سایر مطالب شخص ثالث در این مقاله در مجوز Creative Commons مقاله گنجانده شده است، مگر اینکه در خط اعتباری مطالب به گونه دیگری مشخص شده باشد. اگر مطالب در مجوز Creative Commons مقاله گنجانده نشده است و استفاده مورد نظر شما توسط مقررات قانونی مجاز نیست یا از استفاده مجاز فراتر می رود، باید مستقیماً از دارنده حق چاپ مجوز دریافت کنید. برای مشاهده یک کپی از این مجوز، مراجعه کنیدhttp://creativecommons.org/licenses/by/4.0/ .
